DNA-結合タンパク質
「高濃度条件下でDNA結合タンパク質を1分子直視する」
■背景
DNA組換えは2つのDNAをつなぎ換える機構です。DNA組換えは塩基配列がよく似ているかまったく同じ2つのDNA間で起こります(相同組換え)。傷ついたDNAの修復だけでなく、配偶子(卵や精子)をつくる減数分裂時にも見られ遺伝的多様性を生み出す上でも重要な役割を担っています。ヒトでは、その機能不全は不妊症や乳がんなどの発生と強い関係があることが報告されています。私たちは、DNA結合タンパク質が相同組換えにおいてDNAとどのように相互作用しているのかを明らかにするため、DNA結合タンパク質の1分子直視に取り組みました。
■研究概要
相同組換えの重要な中間体であるホリデイジャンクションDNA(図1)は、2つの相同な二本鎖DNA分子が一本鎖の交差によって連結された特殊な構造をとっています。大腸菌ではRuvA、RuvB、RuvCの3種類のタンパク質がホリデイジャンクションDNAと相互作用して、組換えDNAへと成熟させています。RuvBはRuvAと複合体を形成して、二本鎖DNAを汲み出すことでホリデイジャンクションの移動を促進し、枝分かれの移動のモータータンパク質として機能しています。
私たちは、従来の方法では蛍光1分子イメージングが難しかった高濃度条件下で蛍光1分子イメージングができる直径100 nmのナノ開口アレイを用いて、RuvA-ホリデイジャンクションDNA複合体と相互作用しているRuvB を1分子直視しました(図1、文献1)。このナノ開口アレイはゼロモード導波路と呼ばれ、ガラス基板上のアルミニウム薄膜に形成させたものです。ゼロモード導波路は1分子リアルタイムDNAシーケンシングに応用されています(図1、文献2)。
様々なヌクレオチド条件下で、RuvA-ホリデイジャンクションDNA複合体に結合しているRuvBの分子数を、RuvB に蛍光標識した色素の褪色の段階数から1分子定量したところ、ADPとATPの非加水分解誘導体であるATPγSの存在下では、より安定なRuvA-RuvBホリデイジャンクションDNA複合体が形成されていることがわかりました。これは、ATPがRuvB六量体のリング形成とRuvA-RuvB-ホリデイジャンクションDNA 複合体の形成を相乗的に促進し、ホリデイジャンクションDNA 分岐の移動に重要な役割を果たしていることことを示唆しています。
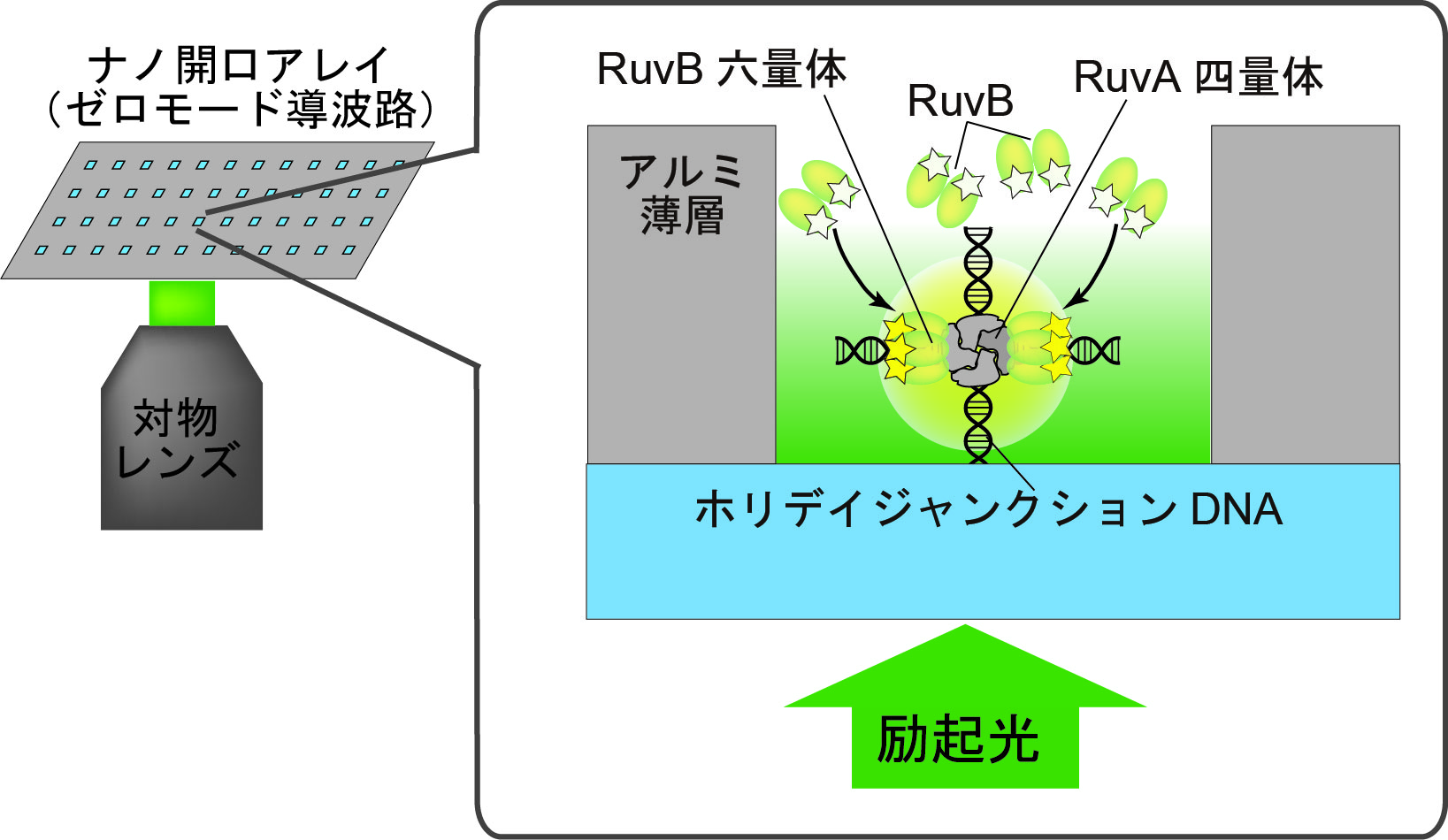
図1 ナノ開口アレイ(ゼロモード導波路)を用いたRuvA-ホリディジャンクションDNAへのRuvBの結合の蛍光1分子イメージングの模式図。
■科学的・社会的意義
従来の蛍光1分子イメージングでは困難であった高濃度条件下での観察は、弱い生体分子間相互作用にかかわる生体分子数の1分子定量や、相互作用の基本的な反応速度論パラメーターの取得を可能にしつつあります。弱い生体分子間相互作用の定量的理解は、タンパク質の機能の謎を解くヒントとなるほか、様々な病態の発現機構や創薬における薬効などの分子レベルでの解明につながります。
■参考文献
1)Iwasa, T., Han, Y. W., Hiramatsu, R., Yokota, H., Nakao, K., Yokokawa, R., Ono, T., Harada, Y. (2015). “Synergistic effect of ATP for RuvA-RuvB-Holliday junction DNA complex formation.” Sci Rep 5: 18177.
2)Eid, J., Fehr, A., Gray, J., Luong, K., Lyle, J., Otto, G., Peluso, P., Rank, D., Korlach, J., Turner, S., et al. (2009). “Real-time DNA sequencing from single polymerase molecules.” Science 323(5910): 133-138.
■良く使用する材料・機器
1) 蛍光顕微鏡 (株式会社ニコン、オリンパス株式会社)
2) 高感度カメラ(アンドール・テクノロジーLtd、浜松ホトニクス株式会社)
3) 光学部品(シグマ光機株式会社、ソーラボジャパン株式会社)
4) 光学フィルター(クロマテクノロジジャパン合同会社、株式会社オプトライン)
5) 防振台(ヘルツ株式会社)
6) 液体クロマトグラフィーシステム(GEヘルスケア・ジャパン株式会社)
7) 遠心機(ベックマン・コールター株式会社)
8) 実験試薬(和光純薬工業株式会社、シグマ アルドリッチ ジャパン合同会社)
 2023年度分野別専門委員
2023年度分野別専門委員
光産業創成大学院大学・バイオフォトニクスデザイン分野
横田浩章 (よこたひろあき)
https://www.gpi.ac.jp/research/bpxd/professor-15/
「DNA結合タンパク質が機能している現場を直視する」
■背景
DNAの遺伝情報をもとにした遺伝子発現、DNA複製、DNA修復、DNA組換えの多くの場面でDNA結合タンパク質が機能しています。これまでこれらの機構に関与する数々のタンパク質の同定と構造が解析され、詳細な生化学的解析に基づいて反応機構のモデルが提唱されてきました。しかしこれらのタンパク質が機能する複合体の3次元構造を解くのが困難なこともあり、タンパク質の相互作用と機能の関係については不明な点が多く残されています。
DNA結合タンパク質の一種であるヘリカーゼは、ヌクレオチドの加水分解エネルギーを用いて二本鎖核酸を一本鎖核酸に巻き戻す酵素です。ヘリカーゼはDNA複製・修復・組換え全ての機構に関与し、原核生物から真核生物まで高度に保存されており、1次構造で高度に保存されている7つのモチーフから6つのスーパーファミリーに分類されています。このうちスーパーファミリー1、2のヘリカーゼはそのほかのスーパーファミリーのヘリカーゼとは異なり非六量体で機能することが知られていました。しかし単量体で機能するのか多量体で機能するのかについて意見が分かれていました。
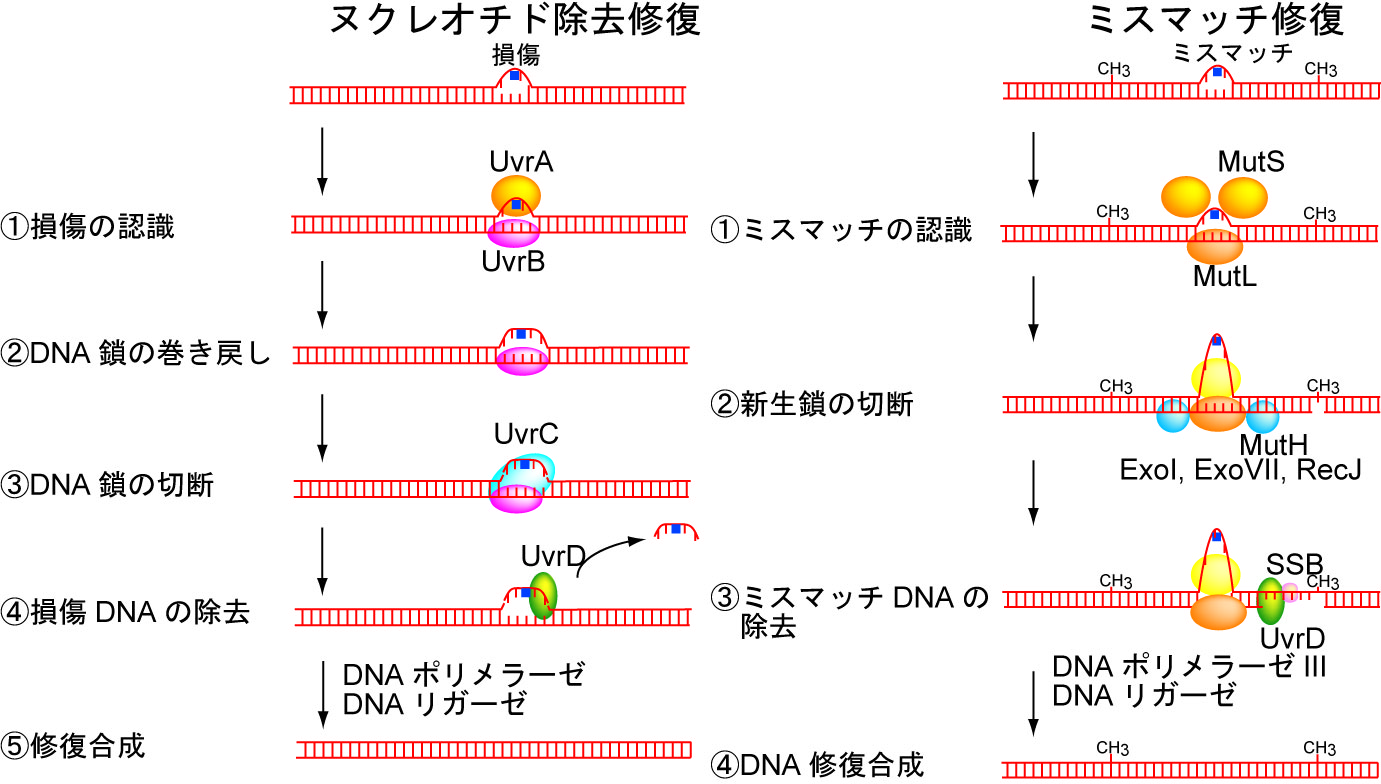
図1 大腸菌のDNA修復機構(ヌクレオチド除去修復・ミスマッチ修復)のモデル図。
■研究概要
そこで大腸菌のDNA修復で損傷やミスマッチ部位を切り出す役割を果たすスーパーファミリー1ヘリカーゼUvrD(図1)に注目し、UvrDを1分子直視することでUvrDが単量体で機能するのか多量体で機能するのかを明らかにしました。UvrDについては、構造生物学的手法を用いた研究からは単量体モデルが(文献1)、生化学的手法を用いた研究からは二量体モデルが提唱されていました(文献2)。UvrDの1分子直視は、蛍光色素Cy5で標識したUvrD1分子を蛍光顕微鏡下で1分子イメージングすることによって行いました。このCy5標識UvrDは遺伝子工学などを用いてUvrD1分子あたりCy5がほぼ1個特異的に標識されるようにしたので、Cy5の蛍光強度の変化を実時間イメージングすることでUvrDのDNA結合分子数を見積もることができます。一方ガラス基板に固定したDNAは蛍光色素Cy3で標識した一本鎖突出二本鎖DNAを用いました。DNAの巻き戻し反応が完了するとCy3で標識されたほうの一本鎖DNAが解離するので、Cy3の蛍光強度の減少によってDNAの巻き戻しを検出することができます。このようにCy5とCy3の2色の蛍光を同時に1分子イメージングしたところ、UvrDは単量体ではなくDNA上で二量体あるいは三量体を形成して効率的にDNAを巻き戻していることを明らかにしました(図2、文献3)。この研究によりUvrDについては多量体モデルが正しいことで決着しました。さらにUvrDが二量体だけでなく三量体でDNA巻き戻しを行っていることが新たに明らかになりました。
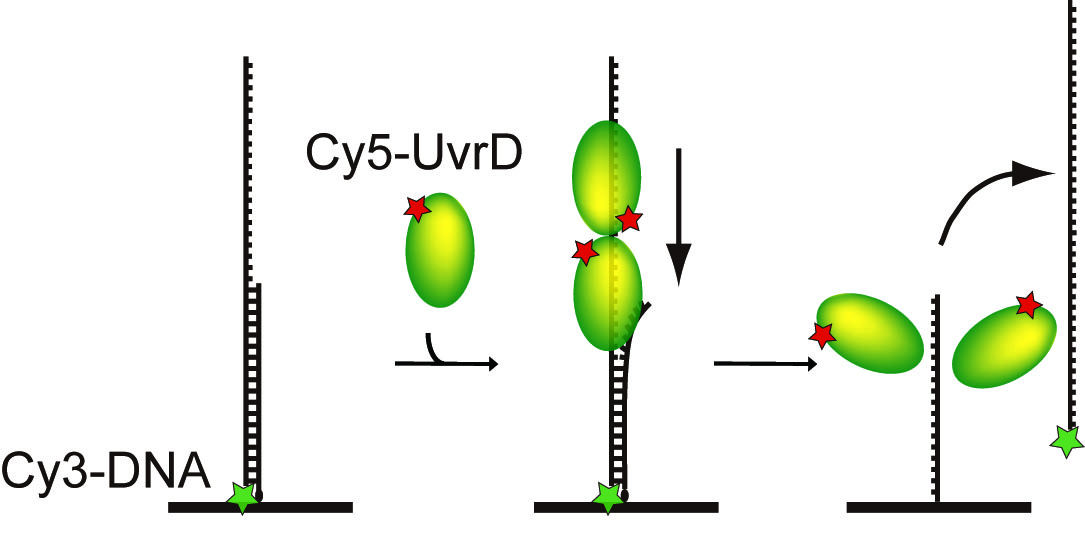
図2 ヘリカーゼUvrDのDNA結合分子数とDNA巻き戻しの蛍光1分子イメージングの模式図。Cy5の蛍光によるUvrDのDNA結合分子数の1分子定量と、Cy3の蛍光によるDNA巻き戻し検出を同時に行った。
■科学的・社会的意義 DNA結合タンパク質の変異などによる機能不全の中にはガンや遺伝病など重篤な病態の発現に関係しているものが多く存在します。生物物理学的手法によってそのダイナミクスを明らかにすることは学術的に意味があるだけでなく、疾病の根本的理解などを通じて医療や創薬などの社会的課題に関係しています。
■参考文献
1)Lee, J. Y., Yang, W. (2006). “UvrD helicase unwinds DNA one base pair at a time by a two-part power stroke.” Cell 127: 1349-1360.
2)Maluf, N. K., et al. (2003). “A dimer of Escherichia coli UvrD is the active form of the helicase in vitro.” J. Mol. Biol. 325: 913-935.
3)Yokota, H., et al. (2013). “Single-molecule imaging of the oligomer formation of the nonhexameric Escherichia coli UvrD helicase.” Biophys. J. 104: 924-933.
■良く使用する材料・機器
1)光学顕微鏡、蛍光顕微鏡(株式会社ニコンインステック、オリンパス株式会社)
2)高感度カメラ(アンドール・テクノロジー社、浜松ホトニクス株式会社)
3)防振台(ヘルツ株式会社)
4)実験試薬(和光純薬工業株式会社)
 H29年度分野別専門委員
H29年度分野別専門委員
光産業創成大学院大学・光バイオ分野
横田浩章 (よこたひろあき)
https://www.sci.osaka-cu.ac.jp/~miyata/index.html
「直接認識と間接認識」
■背景
転写因子などDNA結合タンパク質は特定の塩基配列に結合して遺伝子発現を制御しています。では、DNA結合タンパク質はどのように特定の塩基配列を識別しているのでしょうか?そのメカニズムを明らかにするために、数多くのDNAとタンパク質の複合体の立体構造解析が行われてきました。それらの解析から、アミノ酸残基と塩基の対応にははっきりとした1対1の関係があるわけでないこと、アミノ酸残基と直接相互作用していない塩基配列を変えてもタンパク質とDNAの結合親和性が変わることが分かってきました。では、この特定の塩基配列をのみを認識するメカニズムをどのように理解したらよいのでしょうか?
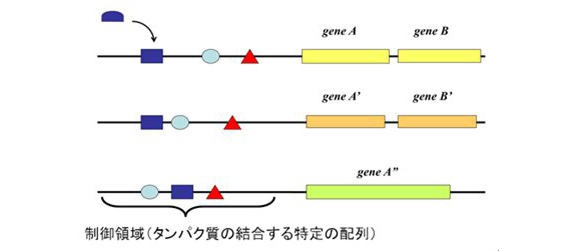
図1 遺伝子の発現は複数のDNA結合タンパク質が制御領域に結合することによってコントロールされている。
■研究概要
この疑問に答えるため、タンパク質立体構造データベース(Protein Data Bank) に登録されたタンパク質とDNAの複合体構造の統計解析(構造バイオインフォマテイィクス解析)を行いました。図2に示すように、DNAの溝にタンパク質が入り込んで、アミノ酸が核酸塩基と直接的に相互作用しているのがわかります。また、DNAはタンパク質が結合することによって曲がっているのが分かります。私たちは、統計的な解析を通して、この2つの異なる性質、つまり、タンパク質とDNAとの直接的な相互作用(直接認識)とDNAの曲がりやすさなどの構造変形のしやすさ(間接認識)の観点からタンパク質のDNA認識機構が説明できることを示しました(文献1)。また、塩基とアミノ酸の相互作用の傾向及び塩基配列ごとのDNAの曲がりやすさを点数化することで、DNA結合タンパク質がどんなDNA配列に結合するか予測することもできるようになりました。さらに、タンパク質の結合する部位についてより詳細なDNA形状の統計解析から、DNAの副溝の幅をタンパク質は認識し分けていること(DNAの形状認識)(文献2)や分子動力学シミュレーション計算によってDNAの曲がりやすさには塩基配列依存性があることなどがわかってきました(文献3)。
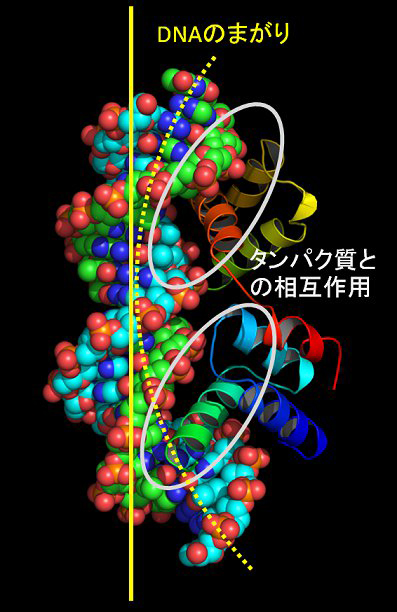
図2 タンパク質とDNAの複合体構造。DNAの溝にタンパク質(らせん状のへリックス構造)の一部が入り込んでいるのが分かる。また、DNAはタンパク質が結合することで曲がっていることが分かる。
■科学的・社会的意義 本研究は、遺伝子の発現制御がどのような分子メカニズムによって制御されているのかを解明するのに役立つ共に、特定のDNAに結合する新たなタンパク質製剤や遺伝子治診断マーカーの開発など様々な応用研究に貢献することが期待されています。
■参考文献
1)Sarai, A. and Kono, H. (2005). “Protein-DNA Recognition Patterns and Predictions” Annu Rev Biophys Biomol Struct 34:379-398.
2)Fujii, S., H. Kono, S. Takenaka, N. Go, and A. Sarai. 2007. Sequence-dependent DNA deformability studied using molecular dynamics simulations. Nucleic Acids Res 35:6063-6074.
3)Rohs, R., Jin, X.,et al. (2010). “Origin of Specificity in Protein-DNA Recoginition” Annu Rev Biochem 79:233-269.
■良く使用する材料・機器
1) PCクラスター
2) スーパーコンピュータ
3) 公共データベース、PDB など
 H27年度分野別専門委員
H27年度分野別専門委員
日本原子力研究開発機構・計算生命情報科学
河野秀俊 (こうのひでとし)
https://yayoi.kansai.jaea.go.jp/rgbf/jp/index.shtml
「RNAポリメラーゼによるDNA転写の分子機構を解明する」
■背景
DNA結合タンパク質のうちでも最も重要で、多くの研究者によって研究されてきたのがDNAの情報をRNAに転写するRNAポリメラーゼです。どのような分子メカニズムでRNAポリメラーゼがDNAを転写するのかを明らかにすることは、生物物理学者にとって大きな課題です。1990年代初めにアメリカの研究グループは非常に巧みな方法で、1分子のRNAポリメラーゼが転写を行う様子を光学顕微鏡を使って観察することに成功しました。
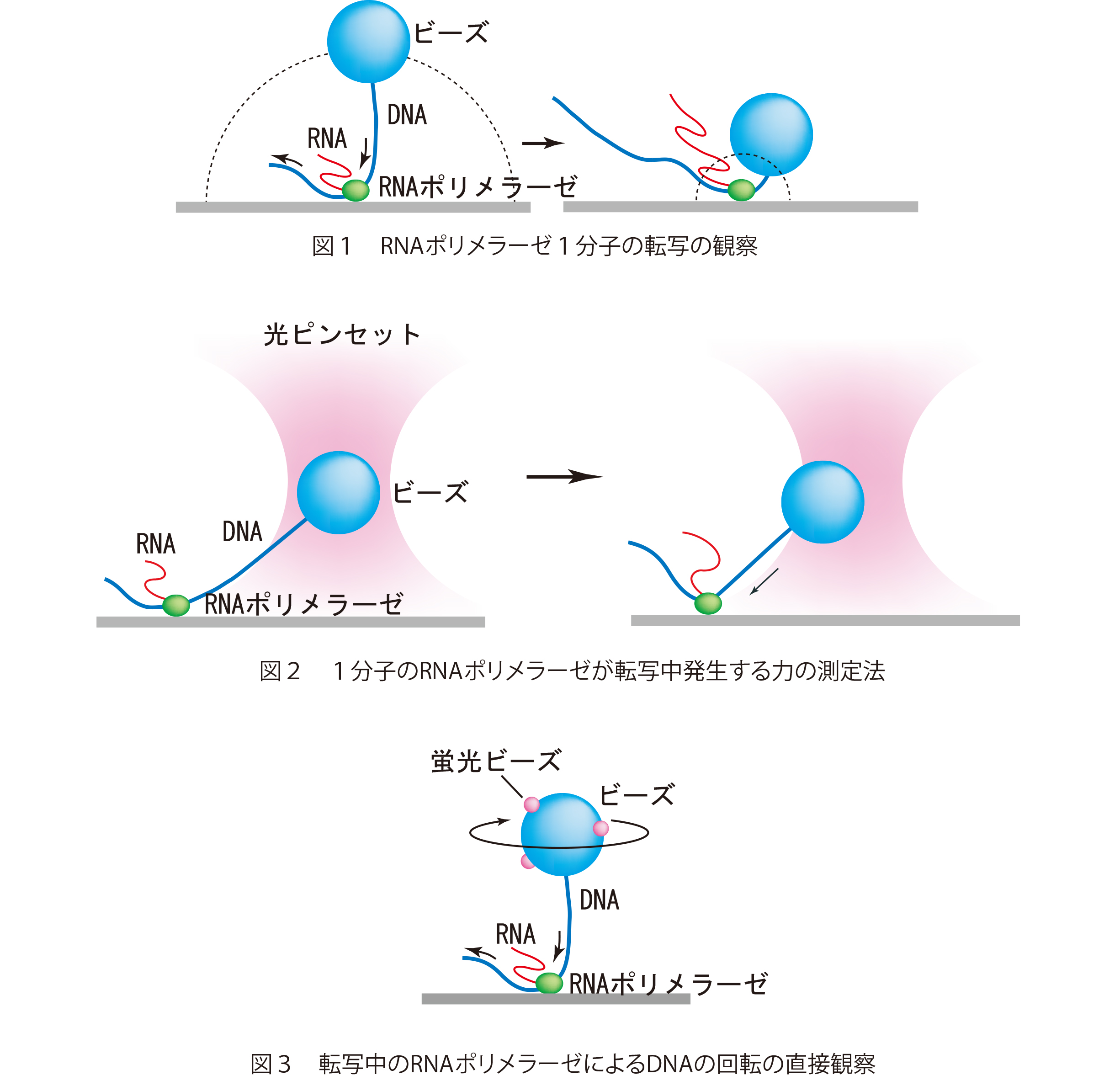
■研究概要
まず、DNAとRNAポリメラーゼからなる転写複合体をつくり、転写を一旦停止させます。この転写複合体を図1のようにRNAポリメラーゼのところでガラス表面に固定します。DNAの片方の端に小さなビーズ(プラスチック製の直径1µm程度の粒子)を結合させます。このビーズを光学顕微鏡で観察すると、ビーズはゆらゆらとブラウン運動しています。ブラウン運動の範囲はポリメラーゼの位置からビーズがくっついているDNAの端までの長さで制限されています。ここで、RNAの材料である4種類の基質ヌクレオチド(ATP、GTP、CTP、UTP)を含む溶液を流し込みます。すると転写複合体の転写反応が再開し、ビーズのブラウン運動が変化していきます。ビーズをDNAの下流(RNAポリメラーゼが転写していく方向の端)に付けた場合、RNAポリメラーゼ分子は転写を進めるにつれDNAをたぐり寄せるため、ビーズのブラウン運動の範囲はしだいに小さくなります(図1)。ビーズの運動範囲の変化からRNAポリメラーゼがたぐり寄せたDNAの長さ、すなわち転写の進み具合を知ることができます。この実験で求められたRNAポリメラーゼの転写の平均速度はおよそ20 塩基/秒で、同じ条件下での溶液中の大腸菌のRNAポリメラーゼの転写速度とほぼ同じ値でした。この方法は、簡単な装置で1分子の酵素が働いている様子を直接観察できる、非常に画期的な方法です。この方法を光ピンセット法*と組み合わせて、ビーズを介してDNAを引っ張った状態で、RNAポリメラーゼによる転写を観察した結果、RNAポリメラーゼ分子は最大25 pNの力でDNAを捕まえていることがわかりました(図2)。また、ビーズに回転を観察するための目印として小さな蛍光ビーズを付けることで、RNAポリメラーゼ分子はDNAを転写する時にDNAの右巻きらせん構造に沿ってDNAを回転させていることが分かりました(図3)。
*光ピンセット法
光を凸レンズで集光したときに、その焦点近傍に屈折率の高い粒子が捕捉される現象があります。この現象を利用して、直径1µm程度のプラスチック製のビーズを捕捉することによって、ビーズに結合させた生体分子を操作する方法を光ピンセット法といいます。
■科学的・社会的意義 生物物理学の研究者は、様々な技術を開発して生体分子の働くメカニズムを研究しています。ここで紹介したRNAポリメラーゼの実験は、その典型的な実験例です。RNAポリメラーゼ以外にも、生体内には様々なDNA結合タンパク質が存在しています。それらのタンパク質についても精力的に研究が行われています。その結果、DNA結合タンパク質分子の働くメカニズムについて、多くのことが分かってきています。
■参考文献
1) Schafer D A, et al.(1991). "Transcription by single molecules of RNA polymerase observed by light microscopy." Nature 352: 444-448.
2) Wang M D, et al. (1998). "Force and velocity measured for single molecules of RNA polymerase." Science 282: 902-907 .
3) Harada Y, et al.(2001). " Direct observation of DNA rotation during transcription by Escherichia coli RNA polymerase. " Nature 409: 113-115.
■良く使用する材料・機器
1)光学顕微鏡、蛍光顕微鏡(株式会社ニコンインステック、オリンパス株式会社)
2)高感度カメラ(アンドール・テクノロジー社、浜松ホトニクス株式会社)
3)防振台(ヘルツ株式会社)
4)実験試薬(和光純薬工業株式会社)
 H26年度分野別専門委員
H26年度分野別専門委員
京都大学 物質-細胞統合システム拠点
原田慶恵(はらだよしえ)
https://www.harada.icems.kyoto-u.ac.jp/index.html
